Introduction J'ai eu l'opportunité de réaliser un stage de 6 semaines au sein
du CHU
d'Amiens, dans le service de
génétique. Ce stage s'est déroulé du 1er juillet au 9 août et a été centré sur la
bioinformatique, un domaine alliant informatique et biologie, essentiel pour le traitement et
l'analyse des données génétiques.
L'objectif principal de ce stage était de paramétrer et d'optimiser une machine Linux pour permettre
aux techniciens de laboratoire de se servir efficacement de pipelines bioinformatiques. Ces
pipelines étaient utilisés pour analyser des séquences d'ADN, en vue de détecter des variants
(mutations génétiques) potentiellement pathogènes. Mon rôle était donc de m'assurer que les
résultats produits par ces analyses soient fiables et exploitables, tout en tenant compte des
spécificités techniques et biologiques liées à ce domaine.
Présentation de l'équipe Le laboratoire de cytogénétique du CHU d’Amiens est
composé de plusieurs services spécialisés qui collaborent étroitement pour assurer un diagnostic
précis et complet. Le service de génétique moléculaire comprend trois techniciens, un biologiste et
un ingénieur, qui se concentrent sur l'analyse des mutations génétiques. Le service d’oncologie et
hématologie est composé de trois techniciens et d’un biologiste, avec un rôle clé dans l'étude des
pathologies sanguines et des cancers. Enfin, le service de caryotype sanguin est formé de deux
techniciens, un biologiste et un ingénieur, spécialisé dans l'analyse des anomalies chromosomiques.
J’ai eu l’opportunité de travailler principalement avec l’équipe de génétique moléculaire, en lien
avec mes missions en bioinformatique. Cependant, la salle où je menais mes activités était un
laboratoire commun, partagé par l’ensemble de ces services. Cela m'a permis de côtoyer
quotidiennement l’ensemble de l’équipe, favorisant des échanges enrichissants et une meilleure
compréhension des différentes disciplines présentes dans le laboratoire.
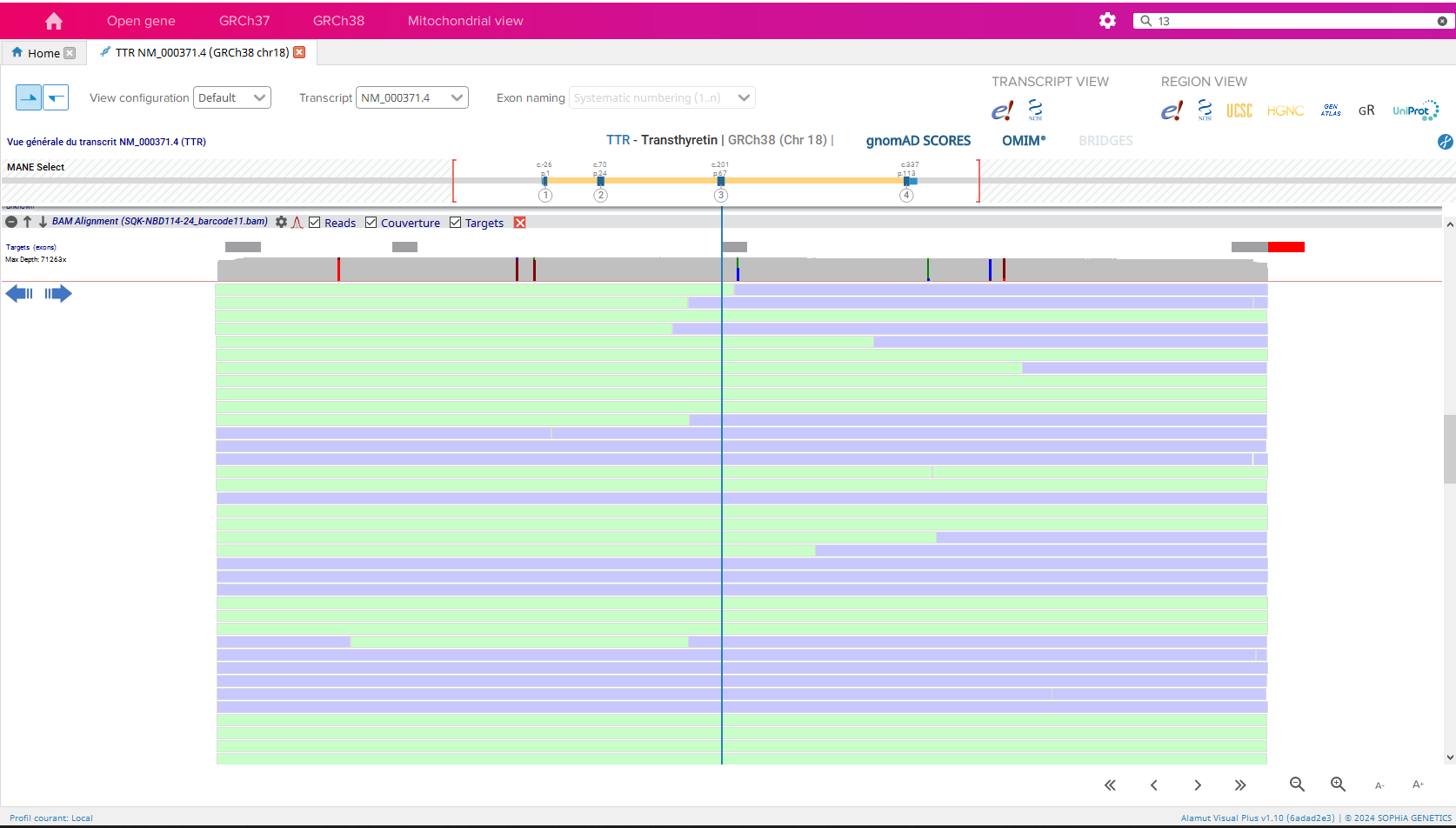
Paramétrage et Optimisation des Pipelines Dès le début de mon stage, ma
mission
principale a été de paramétrer une machine Linux pour permettre aux techniciens du laboratoire de
génétique de séquencer de l'ADN et de détecter des variants génétiques (mutations) potentiellement
pathogènes. Ces techniciens utilisaient une machine de séquençage Nanopore, mais rencontraient des
difficultés avec les résultats produits : les analyses indiquaient la présence de variants à des
emplacements où ils n'auraient pas dû être, rendant les données difficiles, voire impossibles à
exploiter.
Mon travail a consisté à affiner les paramètres du séquençage et de l'analyse des données. J'ai dû
explorer différents modèles de prédiction pour améliorer la fiabilité des résultats. Au départ, il
m'a fallu un certain temps pour m'approprier les notions de génétique nécessaires à la compréhension
des problèmes rencontrés. J'ai donc suivi quelques cours de base en biologie pour être en mesure de
résoudre ces problèmes de manière informatique.
Après deux semaines de travail intensif, j'ai réussi à paramétrer la machine et à fournir des
résultats fiables. Les techniciens ont pu, pour la première fois, obtenir des données exploitables,
et la machine est devenue opérationnelle. Ce succès a marqué une étape cruciale dans mon stage et a
permis de poser les bases pour les missions suivantes.
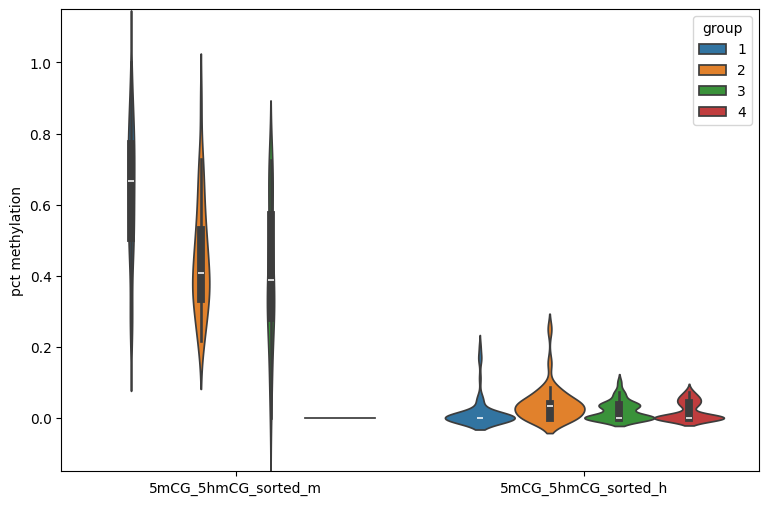
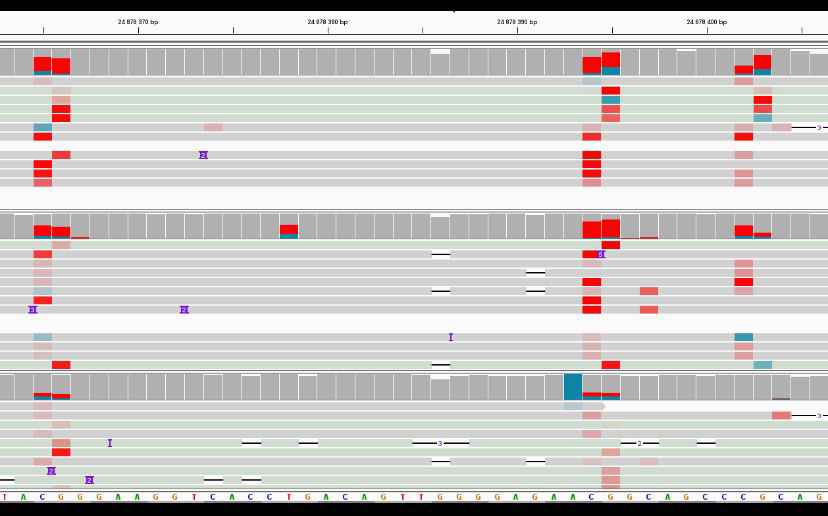
Exploration de la Méthylation ADN Après avoir stabilisé les résultats
du séquençage de
l'ADN, une nouvelle mission m'a été confiée : explorer la méthylation de l'ADN. La méthylation est
un processus biologique complexe, souvent associé à la régulation de l'expression des gènes, et
représente un défi supplémentaire dans l'analyse génétique. Peu étudiée sur l'appareil de séquençage
Nanopore, la méthylation nécessitait une approche nouvelle et innovante.
Le sujet étant extrêmement complexe, et peu documenté sur ce type d'appareil, seulement quelques
équipes dans le monde travaillent activement dessus. J'ai donc dû plonger dans une quantité
significative de littérature scientifique et m'adapter aux spécificités de cet appareil.
Malgré les difficultés rencontrées, j'ai réussi à rendre des résultats exploitables concernant la
méthylation. Ce succès a permis d'ajouter une nouvelle dimension aux capacités analytiques du
laboratoire, ouvrant ainsi la voie à des recherches plus approfondies dans ce domaine crucial de la
biologie.
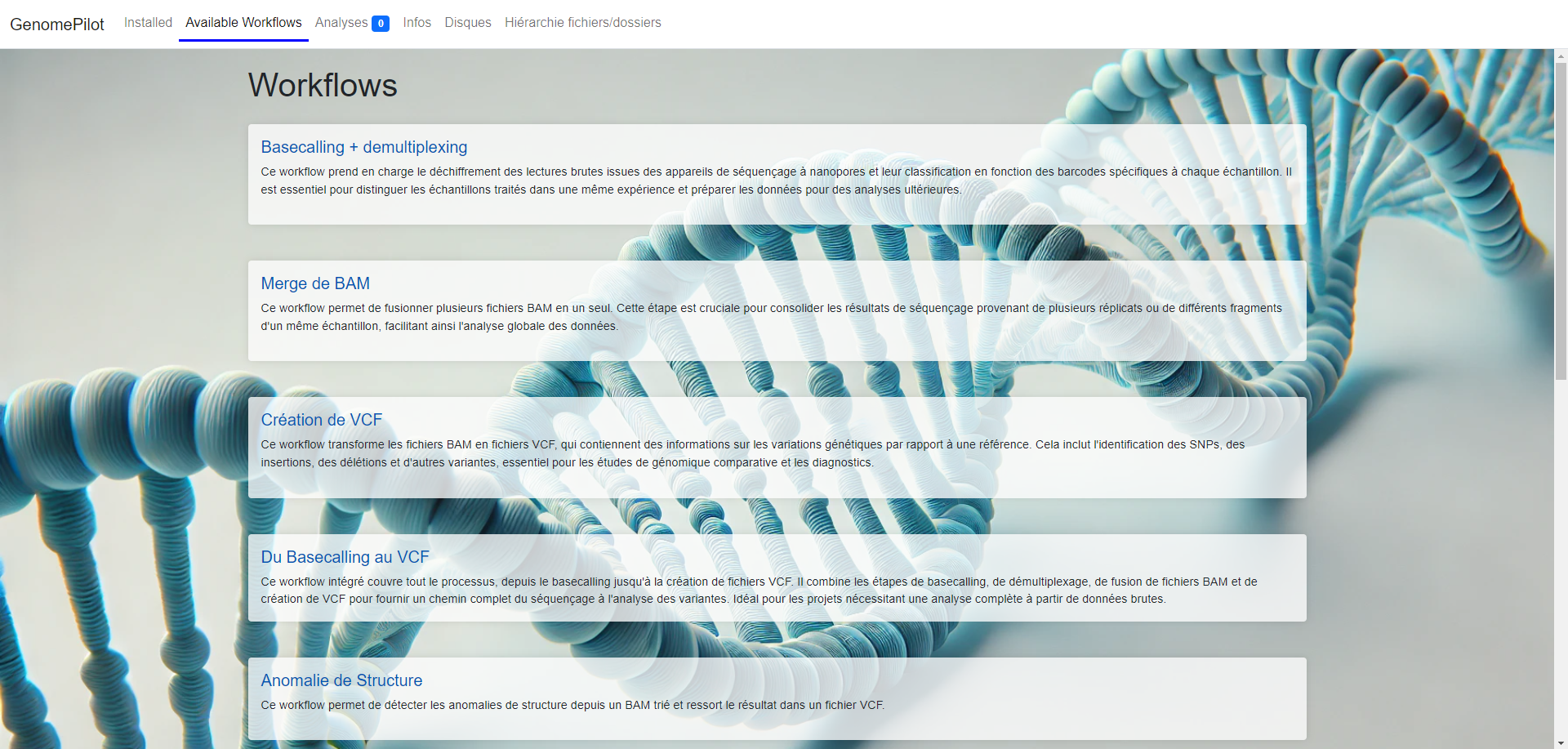
Développement d'une Application Web pour Automatiser les Pipelines À mesure
que mon
stage avançait, il est devenu évident que sans la présence continue d'un bioinformaticien, les
techniciens du laboratoire risquaient de rencontrer des difficultés à maintenir et à exécuter les
pipelines bioinformatiques que j'avais créés. Pour garantir la pérennité de ces processus, j'ai
entrepris de développer une application web qui permettrait de commander et d'exécuter les pipelines
de manière simple et intuitive.
L'idée était de créer une interface utilisateur accessible à tous, même sans connaissances
approfondies en bioinformatique. Chaque pipeline avait sa propre page dédiée, avec des champs
d'entrée clairs pour les fichiers de départ, les références, les dossiers de sortie, etc. De plus,
un panneau de statut permettait de suivre en temps réel l'avancement des tâches : échec, réussite,
ou en cours d'exécution.
L'application intégrait également un système de logs pour suivre l'historique des exécutions et
diagnostiquer les éventuels problèmes. Enfin, j'ai hébergé cette application web en interne au CHU,
garantissant ainsi que les techniciens pourraient continuer à l'utiliser longtemps après la fin de
mon stage.
Bilan et Réflexion sur l'Expérience Mon stage au sein du service de
génétique du CHU
d'Amiens a été une expérience profondément enrichissante sur plusieurs plans. D'un point de vue
technique, il m'a permis d'approfondir mes compétences en bioinformatique et en développement de
pipelines sur des données de séquençage d'ADN. J'ai également appris à naviguer dans un
environnement complexe où la biologie et l'informatique se rencontrent, exigeant une compréhension
fine des deux domaines pour résoudre des problèmes spécifiques.
L'un des plus grands défis que j'ai rencontrés a été d'assimiler les concepts biologiques
fondamentaux pour comprendre les enjeux du séquençage d'ADN et des mutations génétiques. Cette
compréhension était cruciale pour adapter et affiner les pipelines bioinformatiques, garantissant
ainsi des résultats fiables et exploitables par les techniciens et les médecins.
Un autre aspect essentiel de cette expérience a été le développement d'une application web pour
automatiser l'exécution des pipelines. Cette initiative m'a permis de mettre en pratique mes
compétences en développement fullstack et en DevOps, tout en répondant à un besoin concret du
laboratoire. En rendant ces outils accessibles via une interface simple, j'ai pu assurer la
pérennité des processus même après mon départ.
Enfin, ce stage m'a également sensibilisé à l'importance du travail en équipe interdisciplinaire, en
collaborant étroitement avec des techniciens de laboratoire, des médecins et d'autres experts du
domaine. Cette collaboration m'a permis de mieux comprendre les attentes et les besoins des
différents acteurs, et de développer des solutions qui répondent véritablement à leurs exigences.
En conclusion, cette expérience a non seulement enrichi mon bagage technique, mais elle m'a
également offert une meilleure compréhension du domaine médical, de la biologie moléculaire, et de
l'importance des technologies de l'information dans ces secteurs. Je ressors de ce stage avec un
sentiment d'accomplissement, sachant que mon travail a directement contribué à améliorer les
processus de diagnostic et d'analyse au sein du CHU.